“变量重要性图”和变量选择
💡一则或许对你有用的小广告
欢迎加入小哈的星球 ,你将获得:专属的项目实战(已更新的所有项目都能学习) / 1v1 提问 / Java 学习路线 / 学习打卡 / 每月赠书 / 社群讨论
- 新开坑项目:《Spring AI 项目实战》 正在持续爆肝中,基于 Spring AI + Spring Boot 3.x + JDK 21..., 点击查看 ;
- 《从零手撸:仿小红书(微服务架构)》 已完结,基于
Spring Cloud Alibaba + Spring Boot 3.x + JDK 17...,点击查看项目介绍 ;演示链接: http://116.62.199.48:7070 ;- 《从零手撸:前后端分离博客项目(全栈开发)》 2 期已完结,演示链接: http://116.62.199.48/ ;
截止目前, 星球 内专栏累计输出 100w+ 字,讲解图 4013+ 张,还在持续爆肝中.. 后续还会上新更多项目,目标是将 Java 领域典型的项目都整一波,如秒杀系统, 在线商城, IM 即时通讯,权限管理,Spring Cloud Alibaba 微服务等等,已有 3700+ 小伙伴加入学习 ,欢迎点击围观
分类树很好。它们为逻辑回归提供了一个有趣的替代方案。大约七八年前,我开始将它们包括在我的课程中。问题很好(如何获得最佳分区),算法过程很好(根据一个变量拆分的技巧,每个节点只有一个变量,然后向前移动,从不向后移动),以及视觉输出非常完美(具有该树结构)。但预测可能相当糟糕。该算法的性能几乎无法与(明确指定的)逻辑回归竞争。
然后我发现了森林(有关详细介绍,请参阅 Leo Breiman 的页面 )。作为 boostrap 程序的忠实粉丝,我喜欢这个想法。在回归模型中,我通常会提到 bootstrap 以避免渐近近似:我们对行(观察值)进行 bootstrap。就随机森林而言,我不得不承认在每个节点随机选择一组可能变量的想法非常聪明。性能要好得多,但解释通常更困难。而且,当有很多协方差时我喜欢的东西:变量重要性图。这是我们用计量经济学模型很难得到的东西(如果我错了请告诉我)。
为了说明,让我们生成一个大数据集。不一定很大,但是很大,所以我们真的要选择变量。因为如果我们有可能相关的变量会更有趣,我们需要一个协方差矩阵。 R 中有一个很好的包可以随机生成协方差矩阵。
> set.seed(1)
> n=500
> library(clusterGeneration)
> library(mnormt)
> S=genPositiveDefMat("eigen",dim=15)
> S=genPositiveDefMat("unifcorrmat",dim=15)
> X=rmnorm(n,varcov=S$Sigma)
> library(corrplot)
> corrplot(cor(X), order = "hclust")
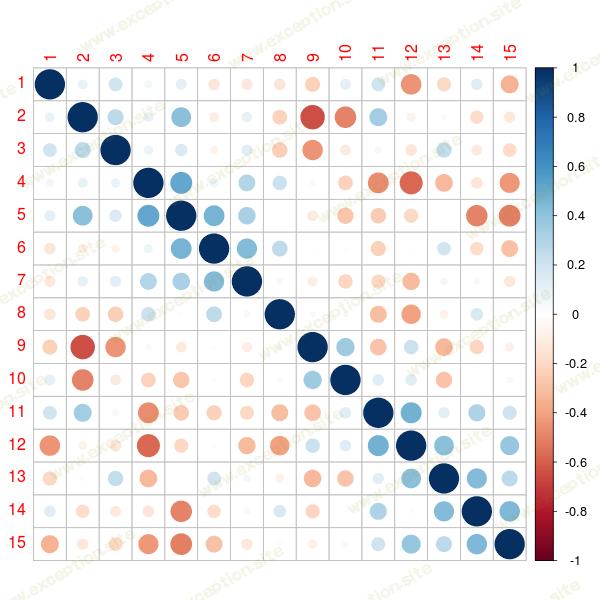
有关该方法的更多详细信息,请参见 Gosh & Hendersen (2003) 。
现在我们有了一个协方差矩阵,让我们生成一个数据集,
> set.seed(1)
> n=500
> library(clusterGeneration)
> library(mnormt)
> S=genPositiveDefMat("eigen",dim=15)
> S=genPositiveDefMat("unifcorrmat",dim=15)
> X=rmnorm(n,varcov=S$Sigma)
> library(corrplot)
> corrplot(cor(X), order = "hclust")
变量重要性图是通过种植一些树获得的,
> set.seed(1)
> n=500
> library(clusterGeneration)
> library(mnormt)
> S=genPositiveDefMat("eigen",dim=15)
> S=genPositiveDefMat("unifcorrmat",dim=15)
> X=rmnorm(n,varcov=S$Sigma)
> library(corrplot)
> corrplot(cor(X), order = "hclust")
然后我们可以使用简单的函数
> set.seed(1)
> n=500
> library(clusterGeneration)
> library(mnormt)
> S=genPositiveDefMat("eigen",dim=15)
> S=genPositiveDefMat("unifcorrmat",dim=15)
> X=rmnorm(n,varcov=S$Sigma)
> library(corrplot)
> corrplot(cor(X), order = "hclust")
也可以使用
> set.seed(1)
> n=500
> library(clusterGeneration)
> library(mnormt)
> S=genPositiveDefMat("eigen",dim=15)
> S=genPositiveDefMat("unifcorrmat",dim=15)
> X=rmnorm(n,varcov=S$Sigma)
> library(corrplot)
> corrplot(cor(X), order = "hclust")
但是我们在所有报道中看到的流行情节通常是
> set.seed(1)
> n=500
> library(clusterGeneration)
> library(mnormt)
> S=genPositiveDefMat("eigen",dim=15)
> S=genPositiveDefMat("unifcorrmat",dim=15)
> X=rmnorm(n,varcov=S$Sigma)
> library(corrplot)
> corrplot(cor(X), order = "hclust")
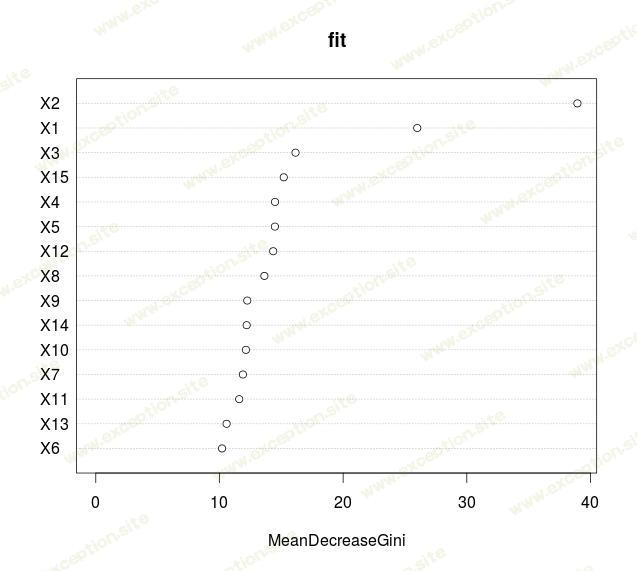
这表示 节点不纯度的平均下降 (而不是 准确性的平均下降 )。正如 Friedman (2001) 所建议的那样,在提升的背景下,还可以可视化部分响应图,
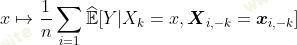
> set.seed(1)
> n=500
> library(clusterGeneration)
> library(mnormt)
> S=genPositiveDefMat("eigen",dim=15)
> S=genPositiveDefMat("unifcorrmat",dim=15)
> X=rmnorm(n,varcov=S$Sigma)
> library(corrplot)
> corrplot(cor(X), order = "hclust")
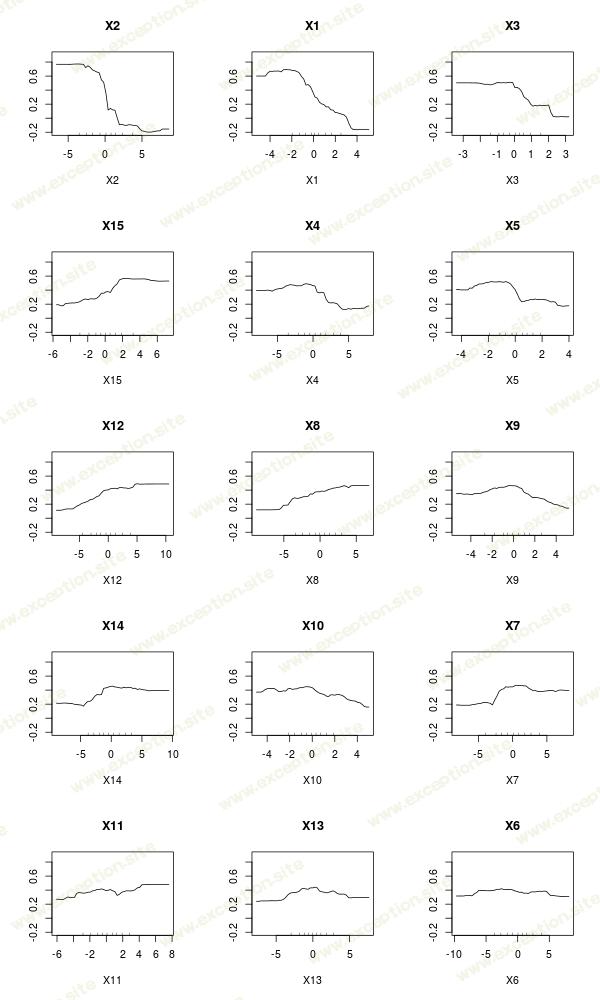
这些变量重要性函数可以在简单的树上获得,不一定是森林。正如
上一篇文章
中所讨论的,给定杂质函数
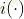 例如基尼指数,如果指数发生变化,我们会在某个节点
t
处进行拆分
例如基尼指数,如果指数发生变化,我们会在某个节点
t
处进行拆分
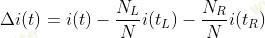
是显着的,其中 tL 是左边的节点, tR 是右边的节点。
在预测 Y 时,可以通过将使用 Xk 的所有节点 t 的 加权杂质减少相加来评估某些变量 Xk 的重要性(对森林中的所有树取平均值,但实际上,我们可以在单棵树上使用它) ,
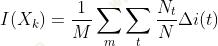
其中第二个总和仅在基于变量 Xk 的节点 t 上。如果 i(.) 是基尼系数,则 I(.) 称为 Mean Decrease Gini 函数。
考虑一棵树,只是为了说明,正如 http://stats.stackexchange.com/ 上的一些旧帖子所建议的那样
> set.seed(1)
> n=500
> library(clusterGeneration)
> library(mnormt)
> S=genPositiveDefMat("eigen",dim=15)
> S=genPositiveDefMat("unifcorrmat",dim=15)
> X=rmnorm(n,varcov=S$Sigma)
> library(corrplot)
> corrplot(cor(X), order = "hclust")
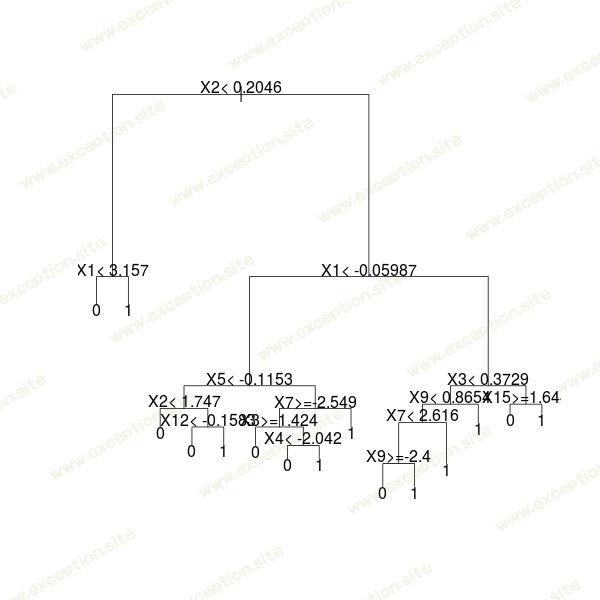
这个想法是查看每个节点哪个变量被用来分割,存储它,然后计算一些平均值(参见 http://stats.stackexchange.com/ )
> set.seed(1)
> n=500
> library(clusterGeneration)
> library(mnormt)
> S=genPositiveDefMat("eigen",dim=15)
> S=genPositiveDefMat("unifcorrmat",dim=15)
> X=rmnorm(n,varcov=S$Sigma)
> library(corrplot)
> corrplot(cor(X), order = "hclust")
这是我们在原始树上得到的变量影响表
> set.seed(1)
> n=500
> library(clusterGeneration)
> library(mnormt)
> S=genPositiveDefMat("eigen",dim=15)
> S=genPositiveDefMat("unifcorrmat",dim=15)
> X=rmnorm(n,varcov=S$Sigma)
> library(corrplot)
> corrplot(cor(X), order = "hclust")
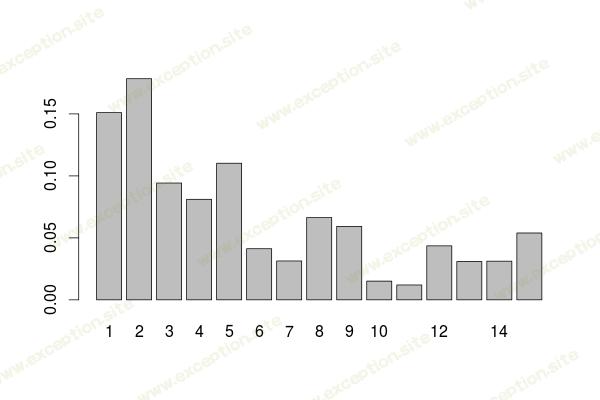
如果我们比较我们在森林里的那个,我们会得到一些非常相似的东西
> set.seed(1)
> n=500
> library(clusterGeneration)
> library(mnormt)
> S=genPositiveDefMat("eigen",dim=15)
> S=genPositiveDefMat("unifcorrmat",dim=15)
> X=rmnorm(n,varcov=S$Sigma)
> library(corrplot)
> corrplot(cor(X), order = "hclust")
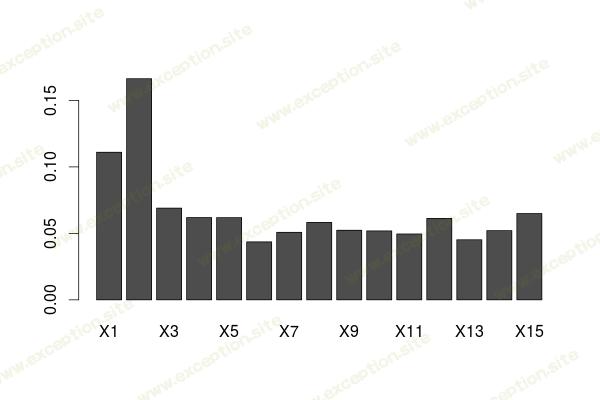
当我们有很多变量时,这张图是一个很好的变量选择工具。如果它足够深,我们可以在一棵树上得到它。
 异常教程
异常教程